- Le Laboratoire
- Médecine de laboratoire
- Logiciel pour séquençage NGS
- SoftGenetics, LLC.

- Produits
- Catalogues
- News & Trends
- Salons
Logiciel pour séquençage NGS NextGENe®de laboratoired'analyse
Ajouter à mes favoris
Ajouter au comparateur
Caractéristiques
- Applications
- pour séquençage NGS, de laboratoire
- Fonction
- d'analyse
Description
Le logiciel NextGENe est le partenaire analytique idéal pour l'analyse des données de séquençage de bureau produites par les systèmes Illumina® iSeq, Miniseq, MiSeq, NextSeq, HiSeq et NovaSeq, les systèmes Ion Torrent Ion GeneStudio S5, PGM et Proton ainsi que d'autres plates-formes.
Le logiciel NextGENe fonctionne sur un système d'exploitation Windows®, qui offre une interface "pointer et cliquer" conviviale pour les biologistes. Il ne nécessite pas de script ou d'autre support bioinformatique, qui sont souvent requis lors de l'utilisation de programmes tels que CLC Genomics Workbench, Lasergene's SeqMan Pro, ainsi que des logiciels académiques tels que MAQ & SOAP, Top Hat, BWA & Bowtie.
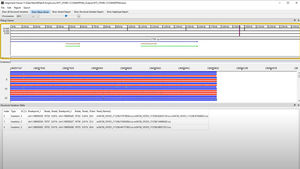
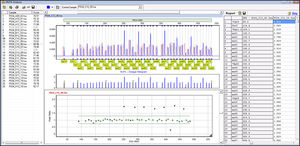
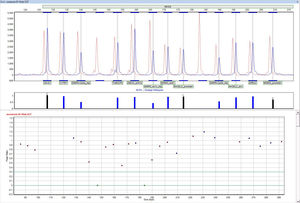
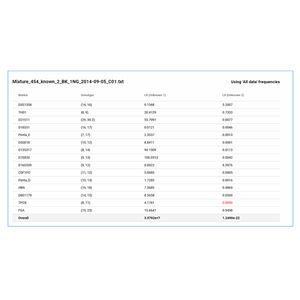
Le logiciel NextGENe utilise des technologies uniques spécifiques aux plates-formes dans un ensemble autonome à applications multiples. Le logiciel NextGENe contient des modules d'analyse pour :
La détection de SNP/Indel
La lignée germinale
Somatique
Capture de l'exome, séquençage de l'exome entier (WES)
Séquençage du génome entier (WGS)
Analyse des variants structurels (y compris la détection des gènes de fusion)
Comparaisons familiales et Trio
Comparaisons tumeur-normal
Détection des variations du nombre de copies (CNV) et alternative au dépistage de type MLPA
assemblage de novo
Transcriptome ; analyse de l'épissage alternatif et niveaux d'expression de la transcription
ChIP-Seq, expression génétique numérique (DGE), analyse et quantification des ARNm et métagénomique
Le logiciel NextGENe est conçu dans un environnement Windows® convivial pour les biologistes, ce qui réduit considérablement le besoin de ressources et de coûts bioinformatiques supplémentaires. Le logiciel NextGENe utilise du matériel 64 bits à faible coût basé sur le système d'exploitation Windows. (Cliquez ici pour une suggestion de configuration matérielle)
---
Recherches associées
- Logiciel viewer
- Logiciel de laboratoire
- Logiciel automatisé
- Logiciel de monitorage
- Logiciel de conception
- Logiciel avec système de suivi
- Logiciel d'importation
- Logiciel serveur
- Logiciel pour la recherche
- Logiciel d'interprétation
- Logiciel de biologie moléculaire
- Logiciel de génétique
- Logiciel pour séquençage NGS
- Logiciel NGS
- Logiciel pour la recherche clinique
- Logiciel pour la génomique
- Logiciel pour séquençage
- Logiciel par électrophorèse capillaire
- Logiciel pour séquençage ADN
* Les prix s'entendent hors taxe, hors frais de livraison, hors droits de douane, et ne comprennent pas l'ensemble des coûts supplémentaires liés aux options d'installation ou de mise en service. Les prix sont donnés à titre indicatif et peuvent évoluer en fonction des pays, des cours des matières premières et des taux de change.