- Produits
- Catalogues
- News & Trends
- Salons
Logiciel pour séquençage NGS GensearchNGSd'analysede gestion de donnéesde diagnostic
Ajouter à mes favoris
Ajouter au comparateur
Caractéristiques
- Fonction
- d'analyse, de gestion de données, de diagnostic
- Applications
- pour séquençage NGS, de laboratoire
Description
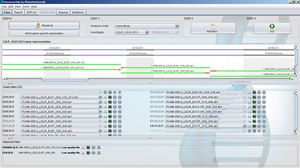
GensearchNGS est une solution logicielle intégrée pour l'analyse des données ADN-Seq des équipements NGS les plus courants tels que Roche/454, Illumina, Ion Torrent et Proton. Il a été conçu et développé en étroite collaboration avec les principaux groupes de diagnostic accrédités en Europe et avec le soutien du projet NMDchip FP7.
Il intègre toutes les étapes depuis l'importation et le filtrage des lectures de séquences, le fractionnement des codes à barres, l'alignement, la détection et l'annotation des variantes jusqu'au rapport final des variantes détectées. Les variantes peuvent être annotées avec diverses sources de données publiques et privées (Ensembl, Alamut, Genome Trax,...). Elles peuvent être filtrées par des combinaisons logiques entre les échantillons pour les études familiales.
Principales caractéristiques :
Logiciel convivial pour le reséquençage des projets des centrales nucléaires dans un cadre de diagnostic, fonctionnant sur les systèmes Windows, Linux et MacOS.
Se connecte*) à des outils d'interprétation de variantes et à des bases de données telles que : Ensembl, dbSNP, Alamut, Clinvar, Wiki Pathways,Gene Ontology*).
Filtrer les variantes d'un échantillon à l'autre (par exemple, les variantes communes à plusieurs échantillons ou exclure les variantes d'un échantillon d'un autre).
Système de base de données centré sur le patient pour gérer les données de séquence et les variantes validées.
Personnalisation possible pour exporter les données dans le système de gestion de votre laboratoire.
Possibilité de publier vos variantes sur Clinvar*).
Développé en collaboration avec des laboratoires accrédités.
Pour les projets plus importants (WES/WGS) : possibilité d'utiliser plusieurs PC en parallèle.
Technologie de plugin : alignement avec nos algorithmes propriétaires ou publics tels que Bowtie, BWA ou Stampy.
Annotation et filtrage avec les descripteurs et codes HPO, OMIM et Orphanet, utiles pour la recherche de mutations dans des exomes ou génomes entiers.
---
* Les prix s'entendent hors taxe, hors frais de livraison, hors droits de douane, et ne comprennent pas l'ensemble des coûts supplémentaires liés aux options d'installation ou de mise en service. Les prix sont donnés à titre indicatif et peuvent évoluer en fonction des pays, des cours des matières premières et des taux de change.