- Produits
- Module logiciel d'analyse
- JSI medical systems

- Produits
- Catalogues
- News & Trends
- Salons
Module logiciel pour séquençage SEQPATIENTd'analysede laboratoire
Ajouter à mes favoris
Ajouter au comparateur
Caractéristiques
- Fonction
- d'analyse
- Applications
- pour séquençage, de laboratoire
Description
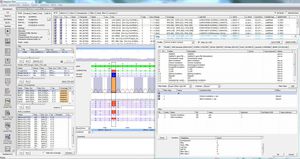
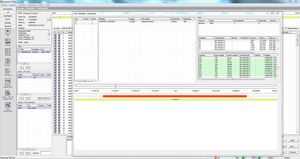
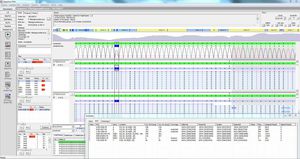
SEQPATIENT est une application puissante et conviviale pour l'alignement et la détection de variantes des données de séquençage Sanger. SEQPATIENT peut analyser des données provenant de toutes les plateformes de séquençage courantes. La visualisation de tous les variants détectés, tels que les délétions, les insertions, les indels et les SNP, est claire et intuitive. L'analyse de l'ADN génomique et de l'ADNc est possible. Une fonction statistique d'aire de crête garantit la détection des mutations à faible fréquence telles que les abandons alléliques, les mosaïques et les variants somatiques.
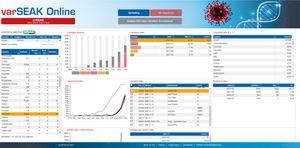
SEQPATIENT offre un accès aux bases de données SNP publiques telles que dbSNP, 1000 Genomes, COSMIC, ClinVar, ClinVitae, ExAC et gnomAD pour la classification et le filtrage. Toutes les données de résultats peuvent être échangées avec notre base de données de variantes pour l'expérience et la connaissance partagées - varSEAK, transférées vers les systèmes LIM internes des laboratoires et / ou émises sous forme de rapports personnalisés pour les patients.
Compatible avec les données de toutes les plateformes de séquençage Sanger courantes
Configuration facile des régions cibles individuelles via l'importation de fichiers txt ou bed basés sur les gènes téléchargés (ENSEMBL / NCBI) ou hg19 / hg38
Appel de base configurable avec des seuils dépendant du séquenceur
Découpage automatique et défini par l'utilisateur des fichiers de résultats de séquençage
Identification des patients et génotypage via les identifiants SNP
Tous les résultats d'un patient sont affichés sur un seul écran, y compris les variants trouvés, les changements d'acides aminés, les nomenclatures HGVS, etc.
Sensibilité et spécificité élevées pour la détection des SNP, des délétions, des insertions et des indels
Base de données de mutations interne conservant l'historique des détections de mutations de SEQPATIENT et SEQNEXT
---
Catalogues
SEQUENCE PILOT
2 Pages
* Les prix s'entendent hors taxe, hors frais de livraison, hors droits de douane, et ne comprennent pas l'ensemble des coûts supplémentaires liés aux options d'installation ou de mise en service. Les prix sont donnés à titre indicatif et peuvent évoluer en fonction des pays, des cours des matières premières et des taux de change.