- Le Laboratoire
- Biologie moléculaire
- Logiciel d'importation
- JSI medical systems

- Produits
- Catalogues
- News & Trends
- Salons
Logiciel de reporting virSEAKd'importationNGSde biologie moléculaire
Ajouter à mes favoris
Ajouter au comparateur
Caractéristiques
- Fonction
- de reporting, d'importation, NGS
- Applications
- de biologie moléculaire
- Type
- automatisé
Description
virSEAK (RUO - research use only) offre une comparaison rapide et pratique de votre séquence SARS-CoV-2 avec les séquences disponibles. Après une configuration personnalisée avec votre kit, il vous suffit de charger des données fastq ou fasta et d'obtenir une séquence alignée avec des variantes.
De plus, votre séquence sera assignée à une lignée de pangolins (par exemple B.1.1.7) et à un clade GISAID (par exemple L).
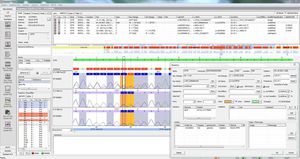
virSEAK vérifie aussi automatiquement si votre séquence contient les variants N501Y et/ou E484K de la protéine spike.
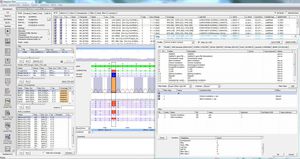
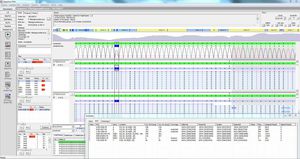
Les valeurs de couverture et de qualité sont affichées, y compris : couverture requise, absolue, moyenne et médiane, % de bases de type "sauvage" et "N". Un indicateur de qualité indique si la qualité satisfait aux seuils de qualité définis.
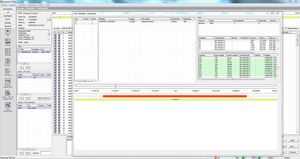
L'outil est également optimisé pour le séquençage NGS à haut débit grâce à des fonctions d'importation et d'exportation automatisées et à un transfert de séquences personnalisable. Il est facile de créer des fichiers multi-fasta pour améliorer la notification à un bureau central de notification pour la surveillance de la situation épidémiologique et de la propagation des mutations (par exemple, pour l'Allemagne, le Robert Koch Institut).
virSEAK - version installable :
importation facile de fichiers de séquençage fastq bruts ou fasta prétraités
séquençage NGS à haut débit avec importation et exportation automatisées par lots
alerte automatique en cas de présence des mutations N501Y et/ou E484K
assignation à une lignée de Pangolins et à un clade GISAID
recherche d'autres variantes avec des fréquences
comparer les sous-types, le nombre et la localisation des résultats, ...
exporter la séquence et les variants (avec les méta-informations, au format fasta et/ou csv)
les résultats sont stockés
séquençage à haut débit entièrement automatisé avec transfert personnalisable des séquences
---
Catalogues
Aucun catalogue n’est disponible pour ce produit.
Voir tous les catalogues de JSI medical systems* Les prix s'entendent hors taxe, hors frais de livraison, hors droits de douane, et ne comprennent pas l'ensemble des coûts supplémentaires liés aux options d'installation ou de mise en service. Les prix sont donnés à titre indicatif et peuvent évoluer en fonction des pays, des cours des matières premières et des taux de change.