- Produits
- Catalogues
- News & Trends
- Salons
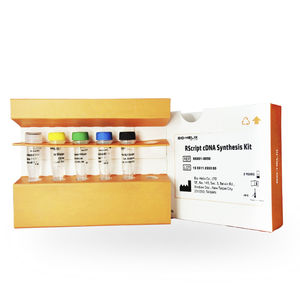
Kit de réactifs transcriptase inverse RScript™colorantpour synthèse d'ADNc
Ajouter à mes favoris
Ajouter au comparateur
Caractéristiques
- Type
- transcriptase inverse
- Applications
- pour synthèse d'ADNc
- Température de stockage
-20 °C
(-4 °F)
Description
Ce kit est développé sur la base de la transcriptase inverse RScript II, qui utilise une amorce de transcription inverse avec une structure en tige-boucle se liant à l'extrémité 3' de la molécule de miRNA. Sous l'action de la transcriptase inverse RScript II, l'ADNc premier brin du miARN artificiellement allongé est obtenu. La séquence générale recommandée pour les boucles souches est 5'-GTCGTATCCAGTGCAGGGTCCGGTATTCGCACTGGATACGAC-3' ; d'autres séquences de boucles souches peuvent également être sélectionnées en fonction des besoins expérimentaux. En règle générale, les amorces de transcription inverse n'ont besoin que d'ajouter 6 bases complémentaires inverses à l'extrémité 3' du miARN à la séquence de la boucle souche. Cette méthode est hautement spécifique et n'effectue la transcription inverse que sur les miARN matures, sans être interférée par ses précurseurs. Les miARN ayant des séquences hautement homologues peuvent être distingués avec précision. Les produits de transcription inverse obtenus avec ce produit peuvent être directement utilisés pour une détection quantitative ultérieure par des méthodes de colorants ou de sondes.
Test de sensibilité du RK004-0050
Le RK004-0050 a été testé avec différentes concentrations (1000 ng, 100 ng, 10 ng, 1 ng, 0,1 ng, 0,01 ng) d'ARN de cellules Hela comme matrice, ciblant le gène has-mir16-5p. Les courbes d'amplification ont montré une sensibilité aussi faible que 10 pg d'ARN total
Performance d'amplification du RK004-0050 sur des modèles provenant de différentes sources (A) ARN de cellules Hela (B) ARN de souris
Le RK004-0050 a été utilisé avec différentes concentrations (1000 ng, 100 ng, 10 ng, 1 ng, 0,1 ng) d'ARN de cellules Hela et d'ARN de souris comme modèles. Les courbes d'amplification pour différents miARN ont montré d'excellentes performances pour Human-U6, has-mir16-5p, has-mir221-3p, Mouse-U6, mmu-mir-5p, mmu-mir24-3p, entre autres.
---
Catalogues
Genomic
6 Pages
Recherches associées
- Kit d'analyse
- Kit de réactifs en solution
- Réactif pour biologie moléculaire
- Kit de réactifs pour la recherche
- Kit de réactifs de protéines
- Kit de réactifs de diagnostic
- Kit de réactifs enzyme
- Kit de test moléculaire
- Kit d'analyse de maladies respiratoires
- Kit d'analyse clinique
- Kit de réactifs milieu réactionnel
- Kit de réactifs colorant
- Kit de réactifs anticorps
- Kit de solution tampon
- Kit d'analyse pour COVID-19
- Kit de réactifs de contrôle qualité
- Kit de réactifs pour PCR
- Kit de réactifs pour bactéries
- Kit d'analyse nasopharyngé
- Kit de réactifs solution de coloration
* Les prix s'entendent hors taxe, hors frais de livraison, hors droits de douane, et ne comprennent pas l'ensemble des coûts supplémentaires liés aux options d'installation ou de mise en service. Les prix sont donnés à titre indicatif et peuvent évoluer en fonction des pays, des cours des matières premières et des taux de change.